学术中心
改进归一化植被指数NDVI估计用于植物健康分类
发布时间:
2024-06-22
来源:
作者:
农业可持续发展对于应对全球粮食安全挑战至关重要。这导致了对植物健康识别自动化的需求。然而,由于缺乏有效的图像处理方法以及需要大量的数据集来训练用于植物疾病诊断的深度学习模型,这一目标受到了阻碍。为了克服对大量训练数据的需求,所提出的局部归一化差异植被指数(Localized Normalized Difference Vegetation Index,LNDVI)使用零样本植物检测模型(Grounded Dino)和最先进的图像分割方法(如分段任意模型(Segment Anything Model,SAM))。这也扩展了系统诊断植物健康的能力,超出了作为训练集一部分的已知植物物种。所提出的系统使用合成归一化差异植被指数(Normalized Difference Vegetation Index,NDVI)单独通过RGB图像来估计植物的叶绿素含量,而不是使用当代作品中使用的RGB和近红外(near Infra-red,nIR)波段的组合。由于NDVI值在很大程度上受到图像捕获时存在的光量的影响,还提出了一种辐射估计度量,该度量使用CIE XYZ(三色激励值)、色调、饱和度和值(Hue, Saturation and Value,HSV)和CIE LAB颜色空间以及相关色温,自动归一化图像健康分类的NDVI阈值,从而能够更精确地分析植物健康。使用Grounding Dino从表型数据集中检测植物的准确率为99.994%。利用并集交集(Intersection over UnionIoU)对图像中的植物区域进行了分割。当使用分段任意模型(SAM)时,集群植物的平均准确度为95.884%,而单个植物的平均精度甚至更好,为97.031%。与NDVI相比,在使用局部归一化植被指数(Localized Normalized Difference Vegetation Index,LNDVI)方法时,观察到植物健康分类存在显著差异。
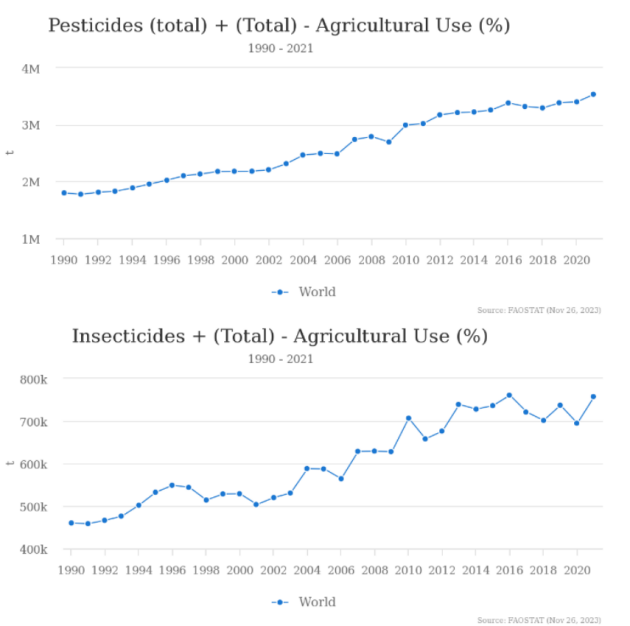 图1 1990年至2020年世界农药和杀虫剂使用情况的可视化。
图1 1990年至2020年世界农药和杀虫剂使用情况的可视化。
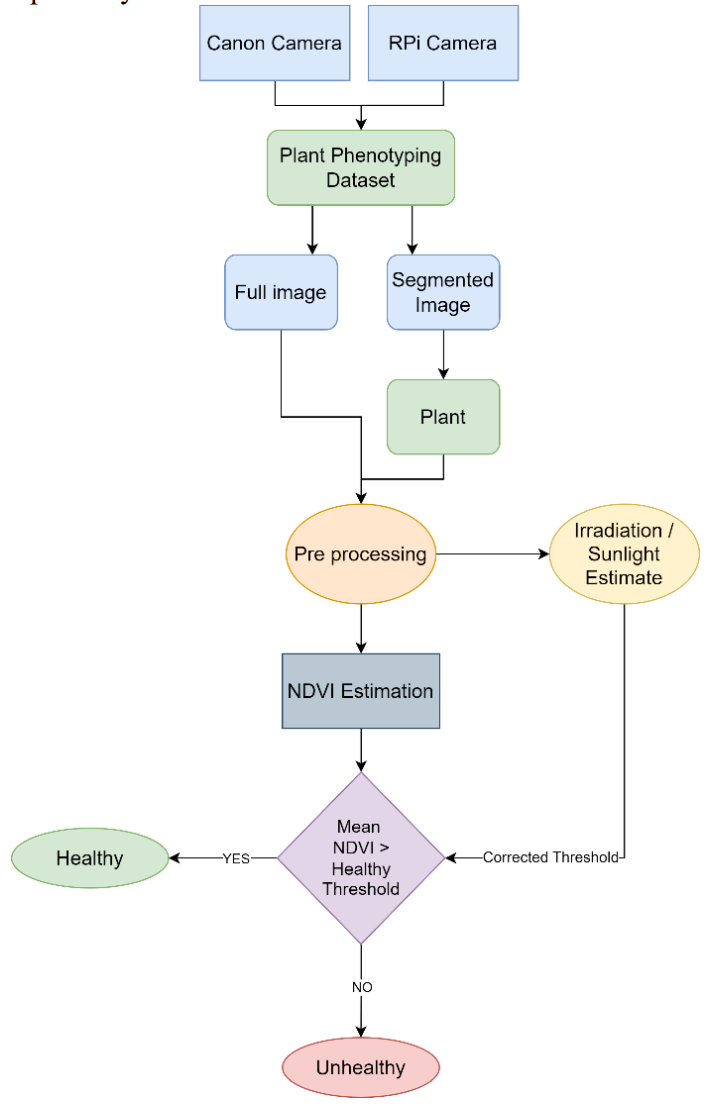 图2 系统的工作流程。
图2 系统的工作流程。
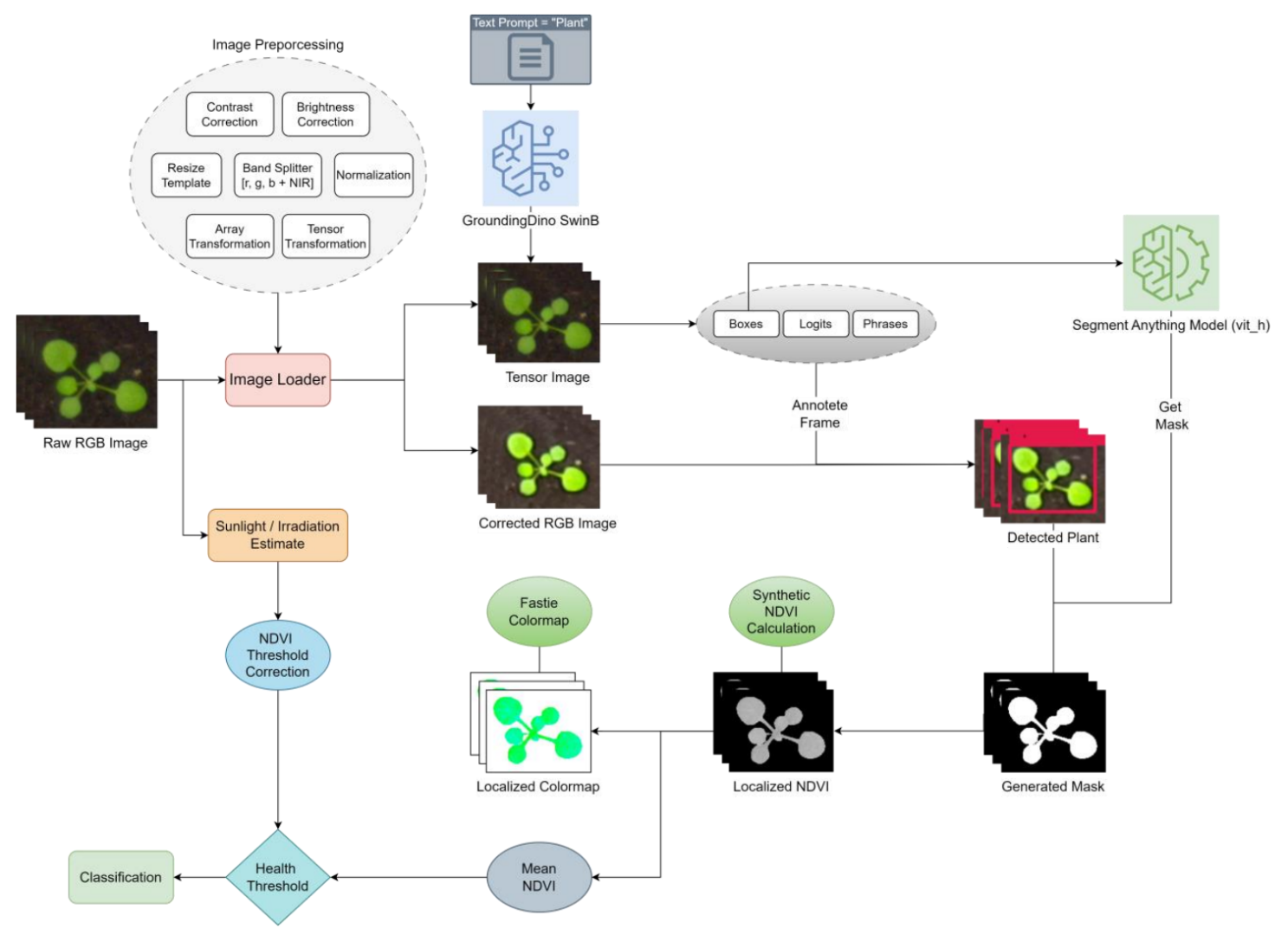 图3 所提出的局部归一化差异植被指数模型的组成部分。
图3 所提出的局部归一化差异植被指数模型的组成部分。
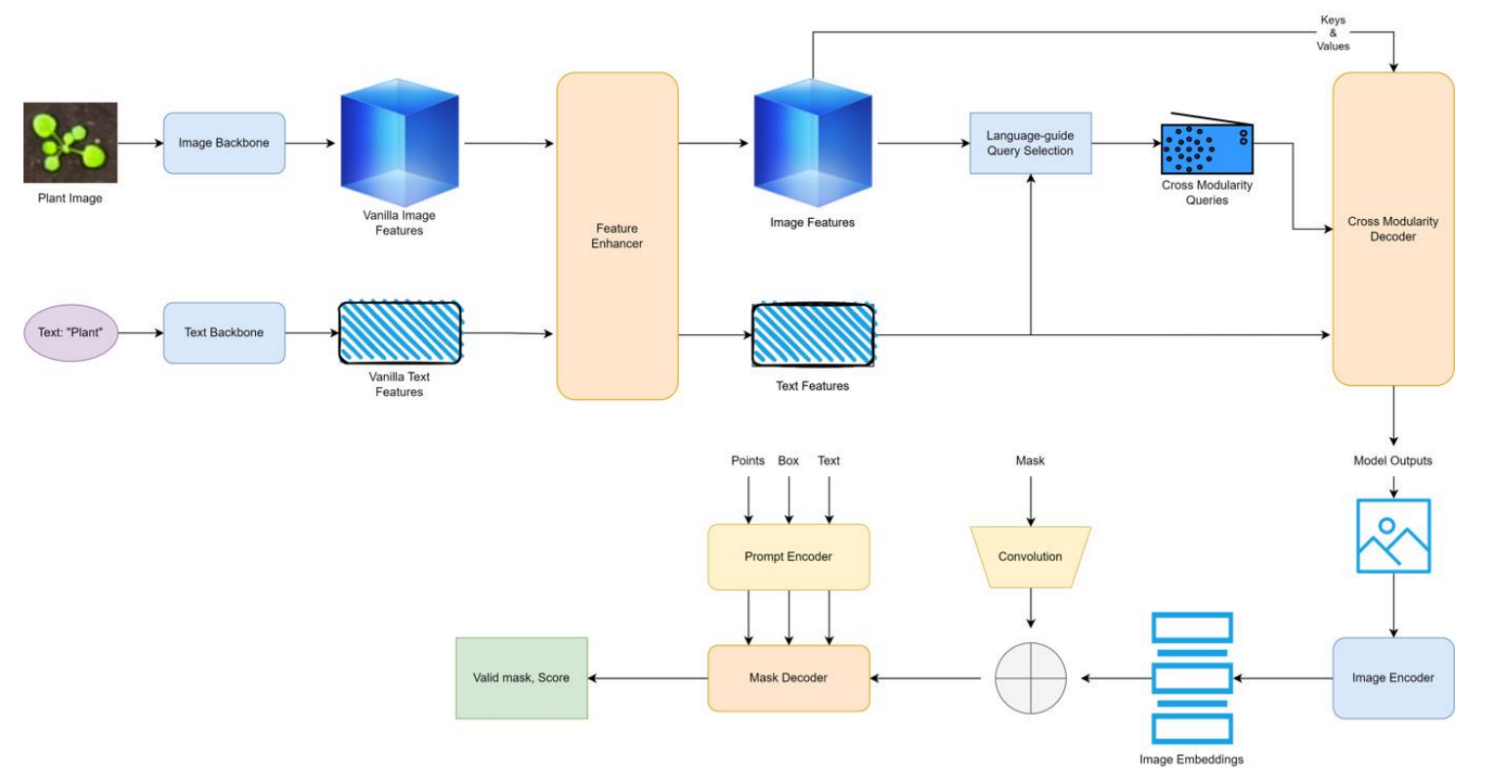 图4 Grounded Dino和SAM的体系结构图。
图4 Grounded Dino和SAM的体系结构图。
 图5 Grounded Dino植物检测。
图5 Grounded Dino植物检测。
 图6 使用SAM的植物分割。
图6 使用SAM的植物分割。
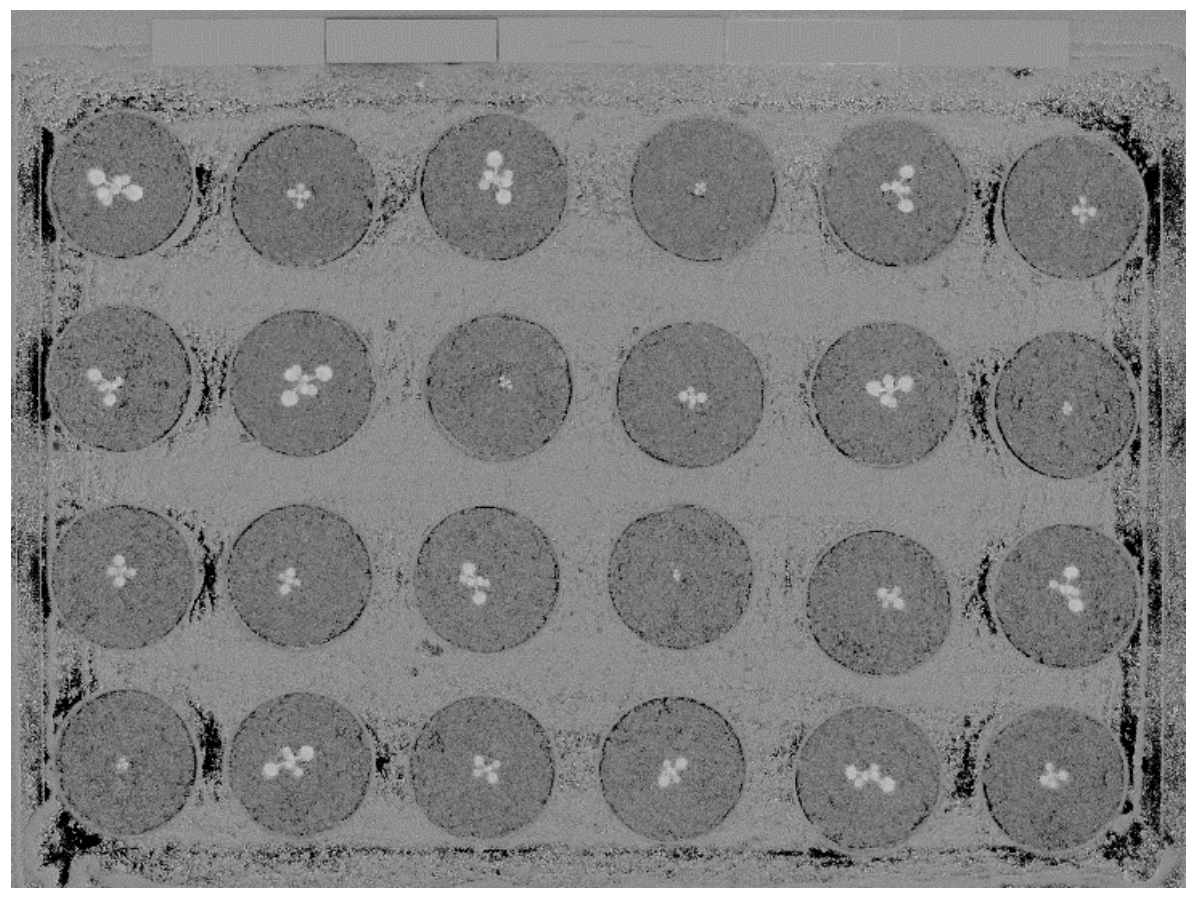 图7 Tray数据集图像的NDVI估计。
图7 Tray数据集图像的NDVI估计。
 图8 Tray数据集图像的彩色NDVI图。
图8 Tray数据集图像的彩色NDVI图。
 图9 Grounded Dino返回的值与真实值的比较。
图9 Grounded Dino返回的值与真实值的比较。
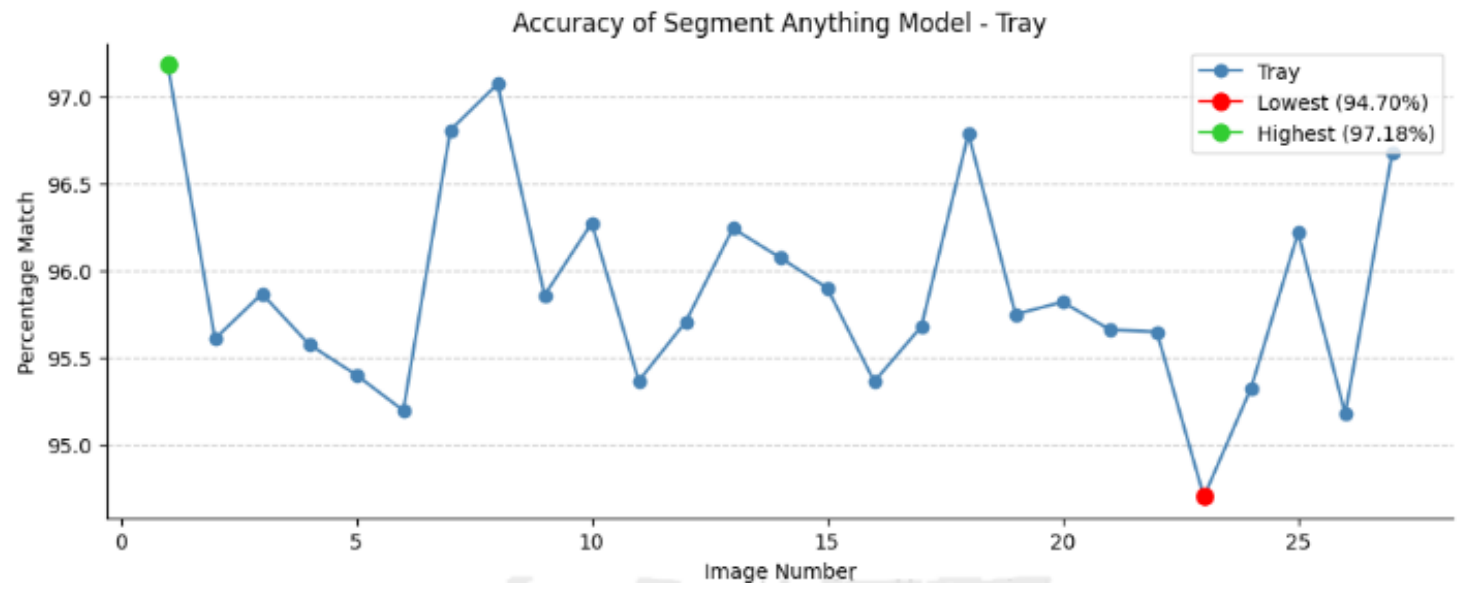 图10 SAM分割的区域百分比与Tray数据集中提供的原始图像片段匹配,平均准确度为95.884%。
图10 SAM分割的区域百分比与Tray数据集中提供的原始图像片段匹配,平均准确度为95.884%。
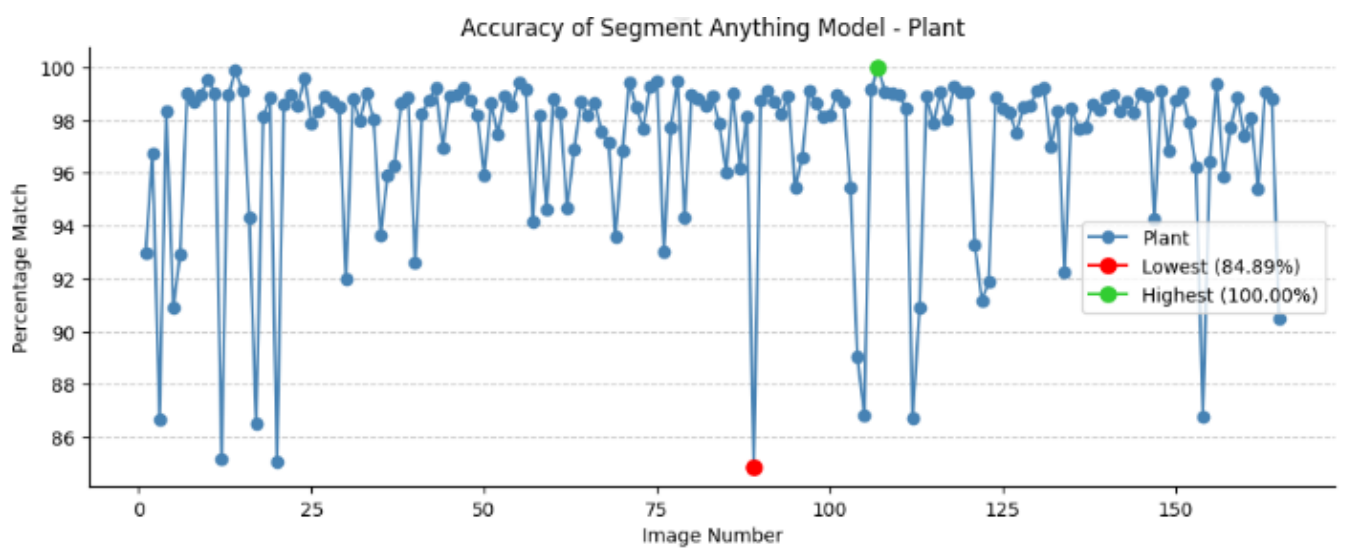 图11 SAM分割的区域百分比与Plant数据集中提供的原始图像片段相匹配,平均准确度为97.031%。
图11 SAM分割的区域百分比与Plant数据集中提供的原始图像片段相匹配,平均准确度为97.031%。
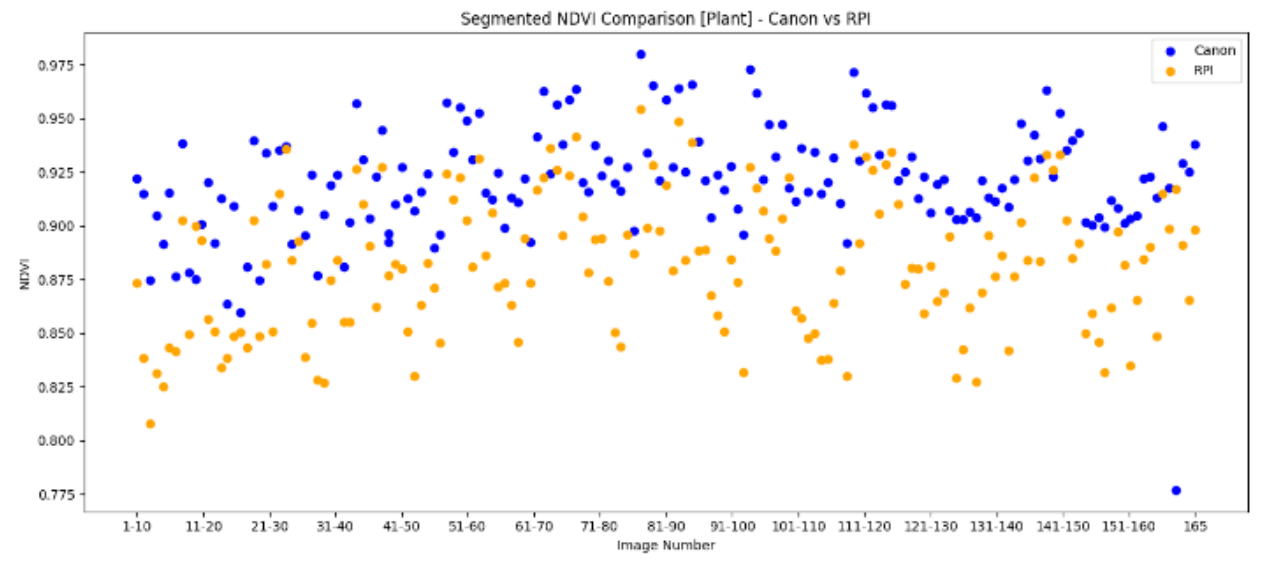 图12 Canon和RPi图像的plant数据集中每个植物的LNDVI值都用不同的颜色表示。
图12 Canon和RPi图像的plant数据集中每个植物的LNDVI值都用不同的颜色表示。
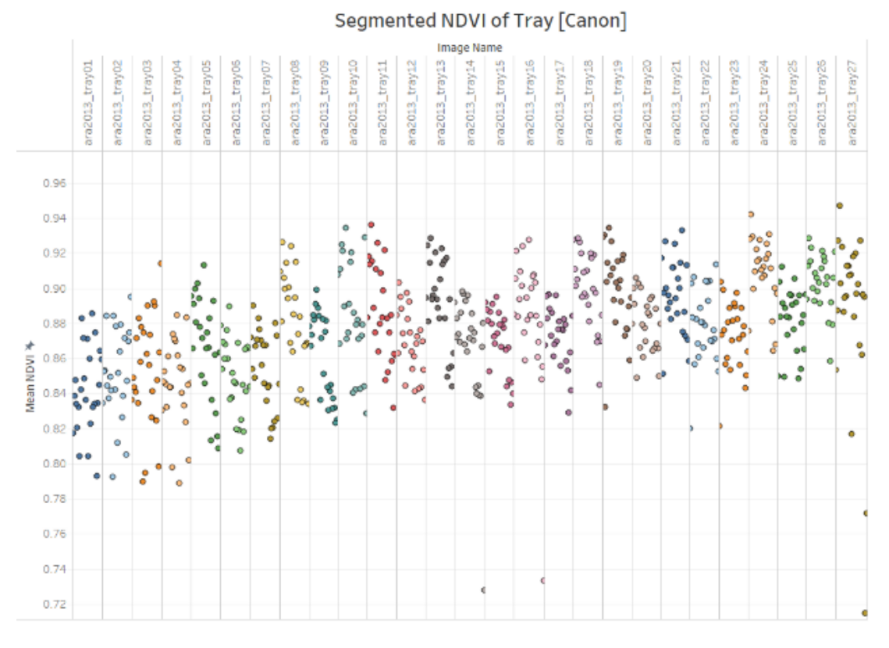 图13 LNDVI为使用佳能相机拍摄的托盘数据集中的每个植物的图像。同一片段中具有相同颜色的圆圈表示同一图像中的植物。
图13 LNDVI为使用佳能相机拍摄的托盘数据集中的每个植物的图像。同一片段中具有相同颜色的圆圈表示同一图像中的植物。
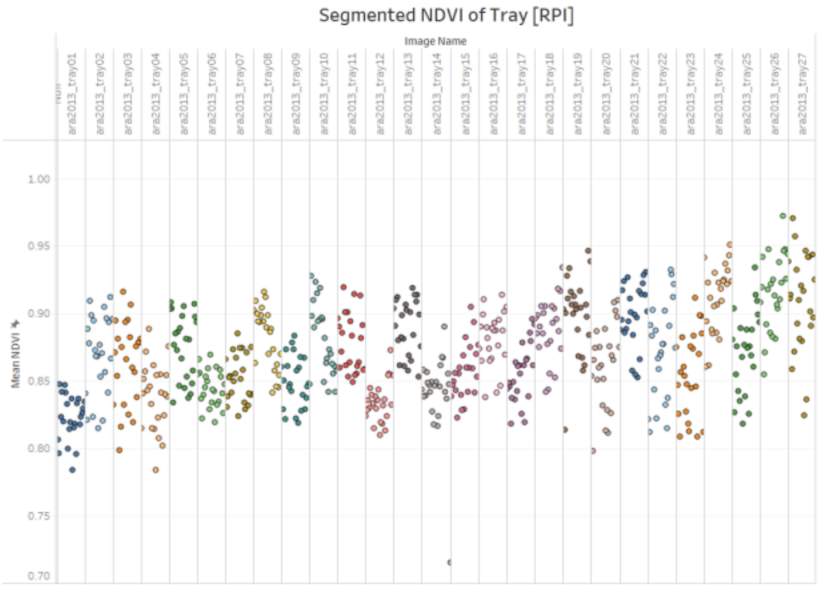 图14 LNDVI——用RPi相机拍摄的托盘数据集中每个植物的图像。相同片段中具有相同颜色的圆圈表示来自相同图像的植物。
图14 LNDVI——用RPi相机拍摄的托盘数据集中每个植物的图像。相同片段中具有相同颜色的圆圈表示来自相同图像的植物。
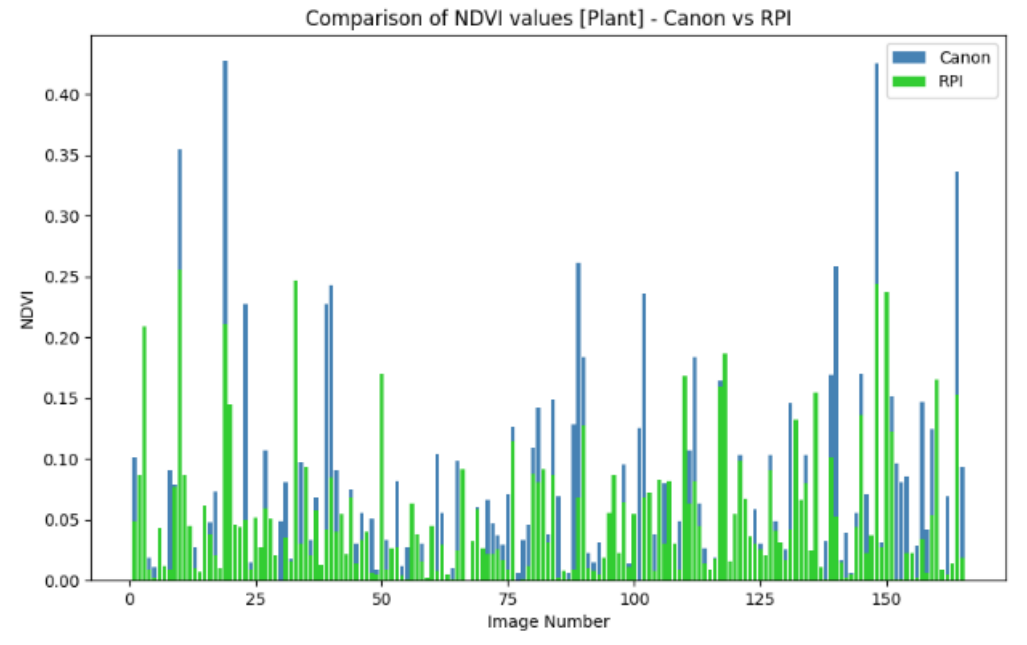 图15 植物数据集RGB图像的合成NDVI值。
图15 植物数据集RGB图像的合成NDVI值。
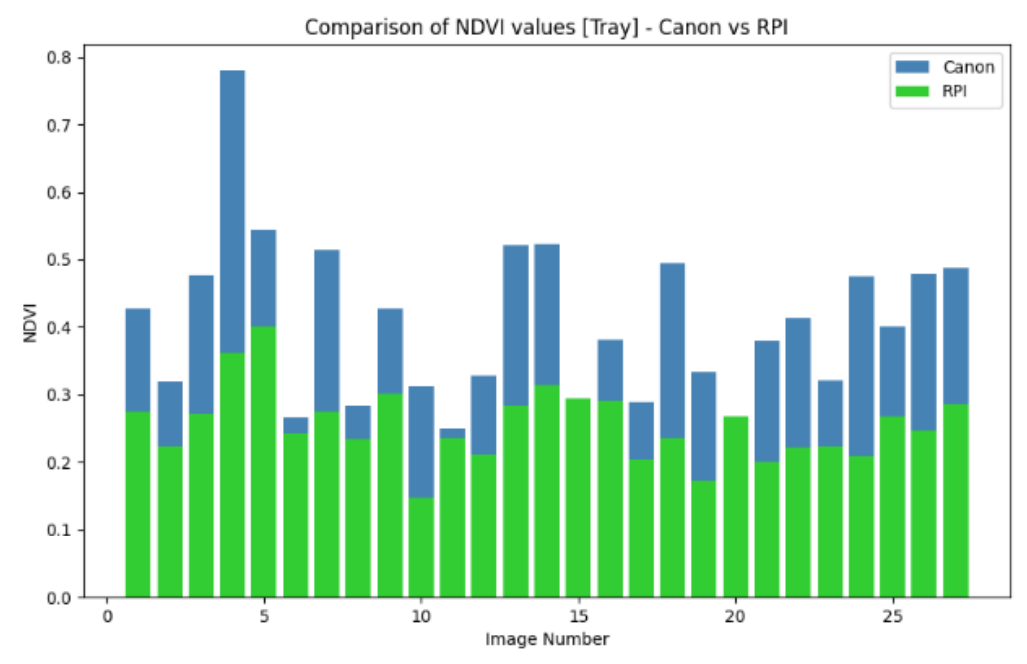 图16 Tray数据集中RGB图像的合成NDVI值。
图16 Tray数据集中RGB图像的合成NDVI值。
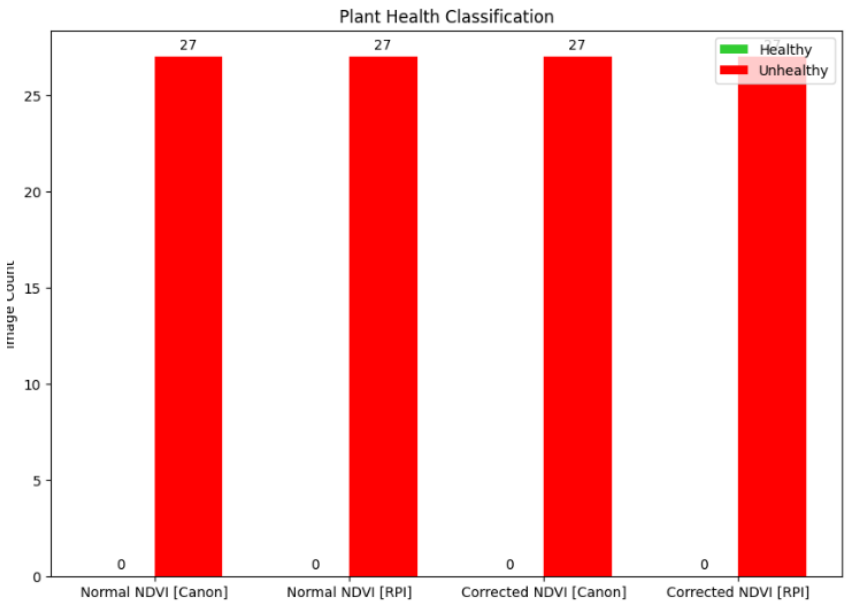 图17 利用Tray图像上的合成NDVI进行植物健康分类的结果。
图17 利用Tray图像上的合成NDVI进行植物健康分类的结果。
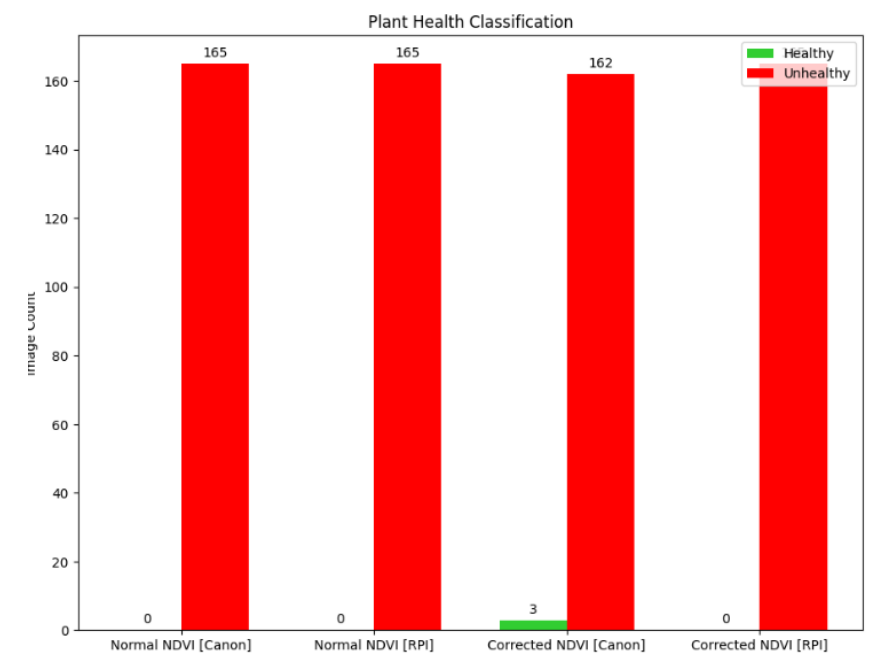 图18 利用合成NDVI对单个植物图像进行植物健康分类的结果。
图18 利用合成NDVI对单个植物图像进行植物健康分类的结果。
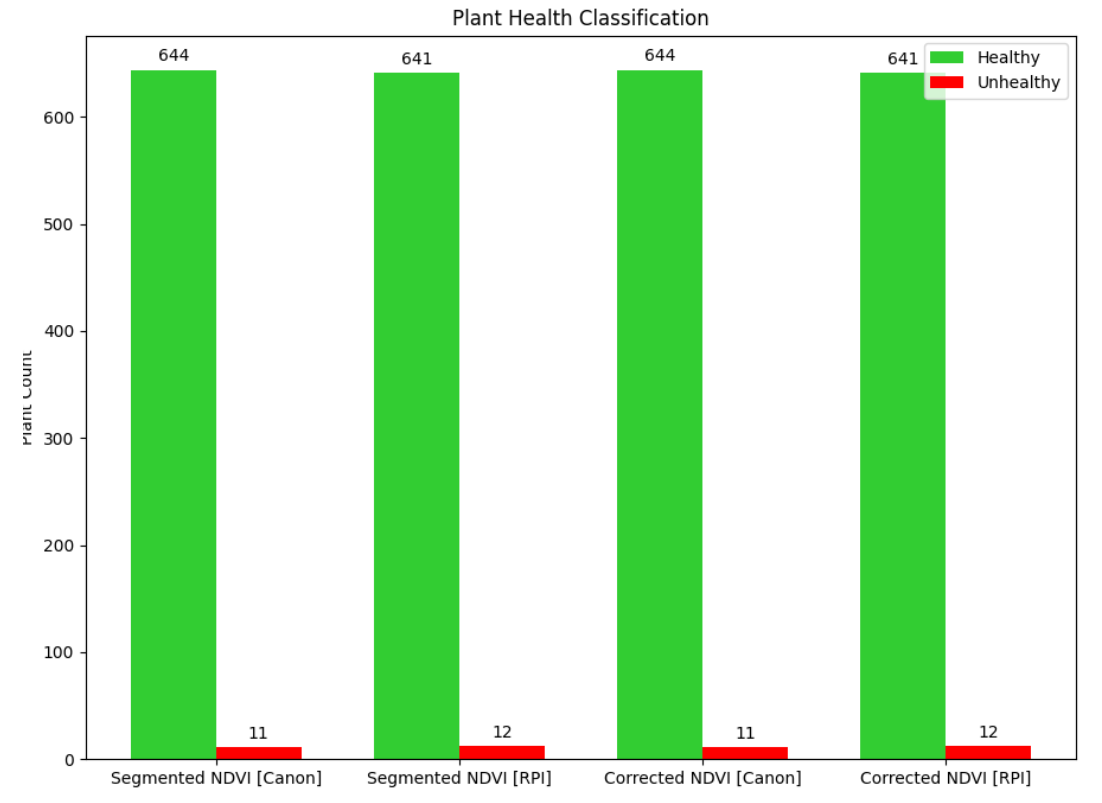 图19 使用LNDVI对Tray图像进行植物健康分类的结果。
图19 使用LNDVI对Tray图像进行植物健康分类的结果。
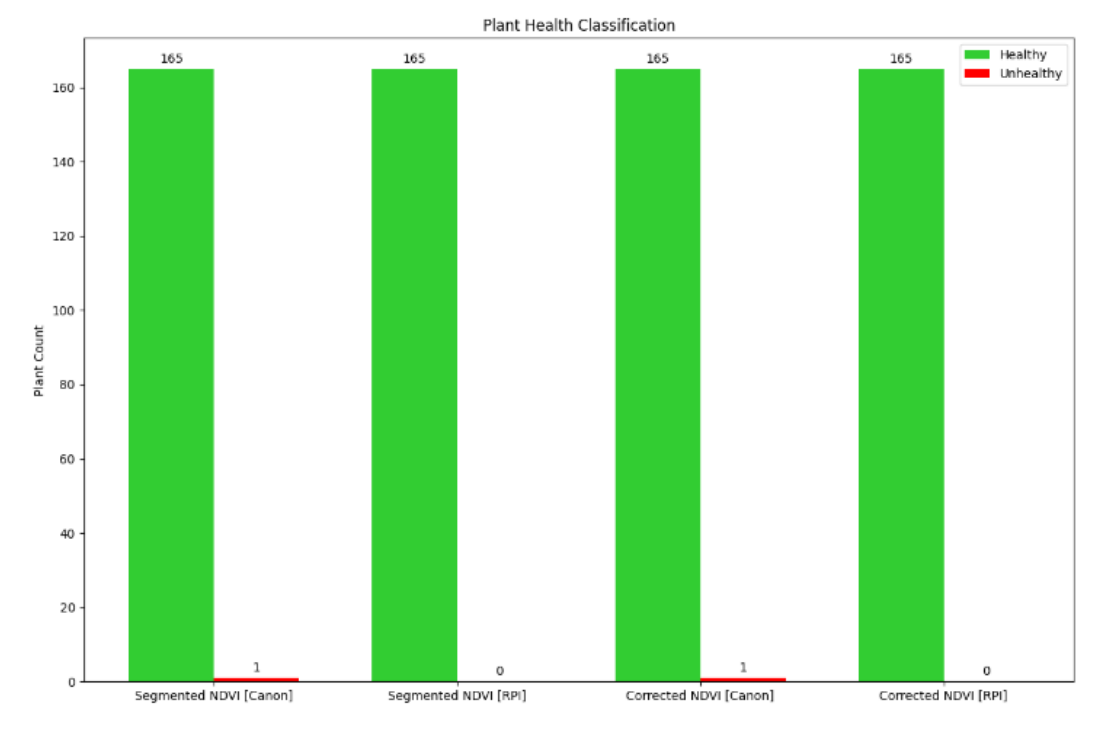 图20 NDVI在植物个体图像上的植物健康分类结果。
图20 NDVI在植物个体图像上的植物健康分类结果。
Ananthakrishnan Balasundaram et al. (2024) An Improved Normalized Difference Vegetation Index (NDVI) Estimation using Grounded Dino and Segment Anything Model for Plant Health Classification. IEEE Access.
编辑
王春颖
推荐新闻
视频展示
