学术中心
中国大豆种质种群两个高通量生理性状QTL等位基因系统的鉴定
发布时间:
2021-03-05
来源:
本站
作者:
PhenoTrait
本文以中国大豆种质种群为试验材料(由341份野生材料(WA),地方品种(LR)和育种品种(RC)组成),研究了与植物生长和产量相关的两个光谱反射生理性状NDVI(归一化植被指数)和CHL(叶绿素指数)的QTL等位基因系统。
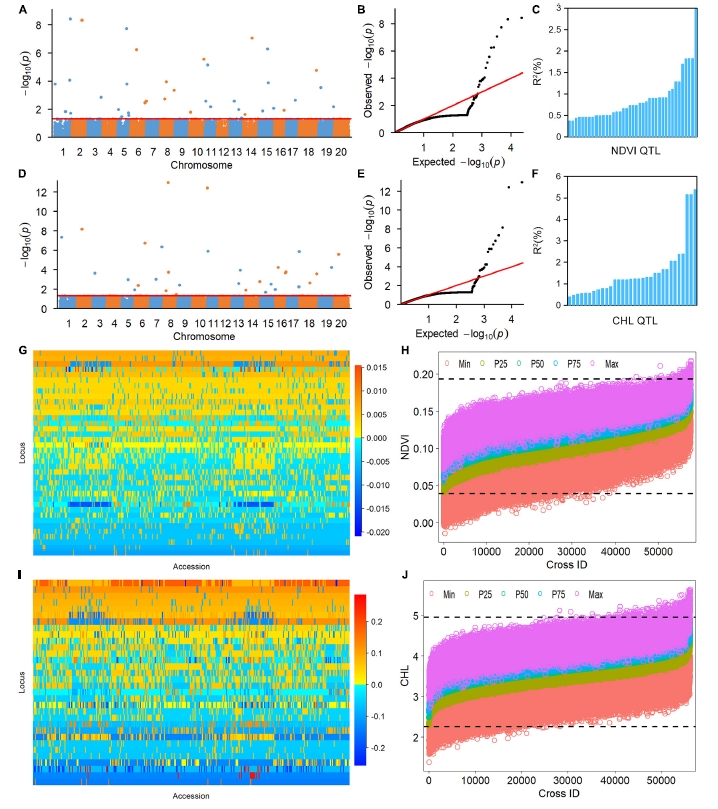
利用RTM-GWAS方法对大豆群体NDVI和CHL表型变异的遗传分析
利用南京农业大学温室高通量植物表型平台的光合成像单元CropReporter进行测量。该平台由传送系统、成像单元(可见光成像、光合表型成像)、灌溉称重单元、控制和分析软件系统、数据库系统等组成。NDVI和CHL数据是从具有两个重复的随机不完全区组设计实验中的多光谱反射图像中获得。NDVI和CHL的范围分别为0.05-0.18和1.20‒4.78,平均值为0.11和3.57,遗传率分别为78.3%和69.2%。LR和RC的NDVI和CHL值均显着高于WA。使用RTM-GWAS(限制性两阶段多位点全基因组关联分析)方法,确定了NDVI和CHL分别具有89和82个等位基因以及每个基因座2-4和2-6个等位基因的38和32个QTL,这分别解释了NDVI和CHL表型变异的48.36%和51.35%。建立了QTL等位基因矩阵,并将其分为WA,LR和RC子群体。从WA到LR + RC,NDVI新生了4个等位基因和2个位点,同时汰除了1个等位基因;CHL新生了6个等位基因,在LR+RC中没有汰除基因。对于NDVI和CHL,分别注释了39个和32个候选基因并进行了GO注释,表明存在复杂的基因网络。NDVI和CHL是上游性状,与下游农艺性状相比,其遗传变化相对保守。本文将高通量表型与RTM-GWAS集成为研究性状的群体遗传学提供了有效参考。
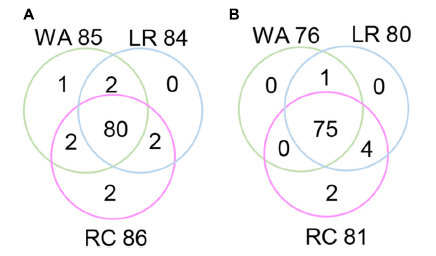
种群间QTL等位基因变化的Venn图
南京农业大学作物表型组学交叉研究中心(PPRC)温室高通量表型平台:
该平台由种植区,灌溉区和PSII成像室组成。使用全自动高通量传输系统将植物从种植区传送到成像室。PSII成像室配备了CropReporter数据采集系统(CropReporter, Phenovation B.V., Netherlands, https://www.phenovation.com/),系统成像面积为70 cm×70 cm,光谱范围为350–1000 nm。在六个不同的波长下捕获光谱反射率图像。从这些图像中,可以估算出每盆植物冠层的NDVI和CHL。该平台配备了实验管理软件IS Agriware Logistics (Version2018.06.99, Indigo Logistics, Netherlands)用来控制系统的运行;CropReporter (Version 4.4.2, Phenovation B.V., Netherlands)控制PSII成像室的CropReporter成像系统,图像分析软件Data Analysis (Version 5.4.8-64b, Phenovation B.V, Netherlands)用来分析PSII成像室采集的数据。所有NDVI和CHL值均直接从平台系统获得。


南京农业大学温室高通量植物表型平台(箭头所指为光合表型成像单元CropReporter)
来源:
Wang L, dong Liu F, Hao X, et al. Identification of the QTL-allele System Underlying Two High-Throughput Physiological Traits in the Chinese Soybean Germplasm Population. Front. Genet., 25 February 2021 | https://doi.org/10.3389/fgene.2021.600444.
推荐新闻
视频展示
